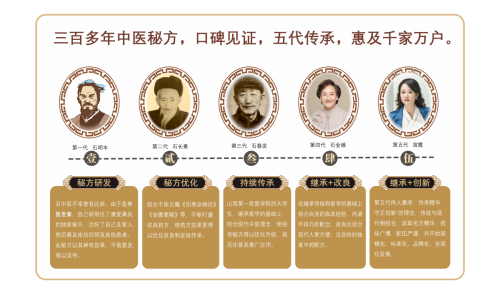
通过连接自连接的三角形M-DNA和矩形M-DNA构造的各种3D多面体的模型和透射电子显微镜(TEM)图像。从左到右:四面体,三角双锥,八面体,五角双锥,三角棱柱,矩形棱柱,五角和六棱柱。
由ASU和上海交通大学(SJTU)组成的一组科学家*,由ASU分子科学学院的Milton Glick教授,ASU生物设计研究院分子设计和仿生中心主任郝浩带领,宣布了这项创新一种新型的元DNA结构,将开拓光电子学(包括信息存储和加密)以及合成生物学的领域。
这项研究发表于2020年9月7日的《自然化学》上—实际上,元DNA自组装概念可能会彻底改变结构DNA纳米技术的微观世界。
众所周知,Watson-Crick碱基对的可预测性质和DNA的结构特征已使DNA可用作设计复杂的纳米级结构和设备的通用构建基块。
Yan解释说:“ DNA技术的里程碑无疑是DNA折纸的发明,其中长的单链DNA(ssDNA)在数百条短的DNA短链的帮助下折叠成指定的形状。”“然而,组装更大(微米至毫米)大小的DNA结构一直是一个挑战,直到最近,DNA结构仍然限制了DNA折纸的使用。”新的微米级结构的长度相当于人类头发的宽度,是原始DNA纳米结构的1000倍。

自2011年以其优雅的DNA折纸纳米结构获得《科学杂志》的封面以来,Yan和合作者一直不懈地努力,利用自然界的灵感,寻求解决复杂的人类问题。
“在当前的研究中,我们开发了一种通用的“元DNA”(M-DNA)策略,该策略允许以亚微米级到微米级大小的各种DNA结构自组装,其方式类似于简单的短DNA链在DNA上自组装的方式。纳米级。严说。
该小组证明,亚微米级的6螺旋束DNA折纸纳米结构(meta-DNA)可以用作单链DNA(ssDNA)的放大类似物,并且两个元DNA包含互补的“ meta-DNA”。碱基对可以形成双螺旋,具有编程的旋律和螺距。
他们使用meta-DNA构件构建了一系列亚微米到微米级的DNA体系结构,包括meta-multi-arm结,3D多面体和各种2D / 3D晶格。他们还证明了在meta-DNA上发生的分层链置换反应,将DNA的动态特征转移到meta-DNA。
在助理教授Petr Sulc(SMS)的帮助下,他们使用了DNA的粗粒度计算模型来模拟双链M-DNA结构并了解所获得的左手和右手结构的不同产量。 。
此外,通过仅改变单个M-DNA的局部柔性及其相互作用,他们就能够构建从一维到3D的一系列亚微米或微米级DNA结构,并具有多种几何形状,包括meta-交界处,超双交叉瓷砖(M-DX),四面体,八面体,棱柱和六种紧密堆积的晶格。
将来,可以使用M-DNA合理设计更复杂的电路,分子电动机和纳米器件,并将其用于与生物传感和分子计算有关的应用中。这项研究将使创建动态微米级DNA结构(在刺激后可重新配置)的可行性大大提高。
作者预计,这种M-DNA策略的引入将使DNA纳米技术从纳米转变为微观规模。这将在亚微米和微米规模上创建一系列复杂的静态和动态结构,从而使许多新应用成为可能。
例如,这些结构可以用作用于构图比先前认为的更大和更复杂的复杂功能组件的支架。这一发现也可能导致更复杂和复杂的行为,这些行为通过结合不同的基于M-DNA的分层链置换反应来模拟细胞或细胞成分。
参考:姚光宝,张菲,王菲,彭天焕,刘浩,埃里克·波普尔顿,彼得·舒尔茨,姜硕行,刘岚,陈恭,荆欣欣,刘晓国,王丽华,刘艳,春海的“元DNA结构”范和焰,2020年9月7日,自然化学。
10.1038 / s41557-020-0539-8
* ASU团队成员:姚光宝(共同第一作者,张飞(现任罗格斯大学助理教授,共同第一作者),刘浩,埃里克·波普尔顿,彼得·舒尔克,姜硕兴,刘岚,陈恭,刘彦,闫浩(合著者) )
上海交大的团队成员:王菲(第一作者),彭天焕,景新新,刘晓国,王丽华,范春海(共同通讯)
 健康教育网
健康教育网