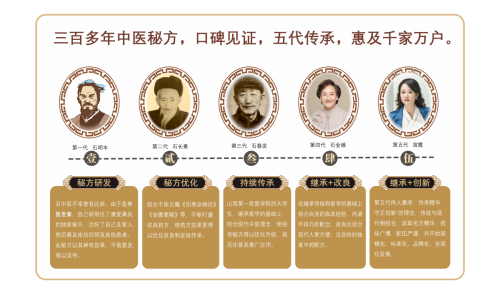
麻省理工学院生物学工程师已经设计了这里示出的存储器存储系统,作为DNA嵌入式仪表,该仪表正在记录人体细胞中的信号通路的活动。
新出版的研究详细介绍了生物工程师如何从麻省理工学院制定一种方法来记录人体细胞DNA中的复杂历史,使它们通过测序DNA来检索过去事件的“存储器”。
这个模拟存储器存储系统 - 首先,可以记录人体细胞中事件的持续时间和/或强度 - 也可以帮助科学家研究细胞在胚胎发育过程中如何分为各种组织,细胞如何经历环境条件,以及它们如何接受遗传变化导致疾病。
“为了能够更深入地了解生物学,我们设计了能够根据基于遗传编码记录仪报告自己历史的人类细胞,”电气工程与计算机科学副教授和生物工程副教授蒂莫西·鲁他补充说,这项技术应对如何对细胞内的基因调节和其他事件产生有助于疾病和发展的洞察力。
Lu是MIT Chinese of Electronics的MIT Synthetic Biology Group负责人,是新研究的高级作者,它出现在科学的在线版本中。本文的牵头作者是Samuel Perli SM '10,PHD '15和研究生Cheryl Cui。
模拟内存
许多科学家(包括LU)都设计了在活细胞中记录数字信息的方法。使用称为重组酶的酶,它们在发生特定事件时将细胞移开其DNA的切片,例如暴露于特定化学品。但是,该方法只显示事件是否发生,而不是有多少曝光或持续多久。
LU和其他研究人员以前设计了如何在细菌中记录这种类似物信息的方法,但到目前为止,没有人在人体细胞中达到它。
新的方法方法基于已知为CRISPR的基因组编辑系统,其由称为CAS9的DNA切割酶和短的RNA链,其将酶引导到基因组的特定区域,引导CAS9在其中切割的地方。
CRISPR广泛用于基因编辑,但麻省理工学院团队决定适应内存存储。在细菌中,在Crispr最初进化的地方,系统记录了过去的病毒感染,使细胞可以识别和争取入侵病毒。
“我们希望调整CRISPR系统来存储人类基因组中的信息,”Perli说。
在使用CRISPR以编辑基因时,研究人员会产生与宿主生物体基因组中靶向序列相匹配的RNA引导股。为了编码记忆,麻省理工学院队采用了不同的方法:它们设计了识别编码相同引导股的DNA的导向股,从而创建他们称之为“自定位指南RNA”。
通过该自定位引导RNA链的LED,CAS9切割编码引导股的DNA,产生成为事件永久记录的突变。一旦突变,DNA序列产生新的引导RNA链,其将CAS9指向新突变的DNA,只要CAS9是活性的,只要表达自靶向导向RNA即可累积即可积累。
通过使用传感器用于调节CAS9或自定位指导RNA活性的特定生物事件,该系统使得作为这些生物输入的函数积累的渐进突变,从而提供基因组编码的记忆。
例如,研究人员设计了一种基因电路,该基因电路仅在靶分子的存在下仅表达CAS9,例如TNF-α,其通过免疫细胞在炎症期间产生。每当存在TNF-α时,CAS9切割编码引导序列的DNA,产生突变。暴露于TNF-α或越大的TNF-α浓度越长,在DNA序列中越突出。
通过稍后测序DNA,研究人员可以确定有多少曝光。
“这是我们正在寻找的丰富的模拟行为,在那里,随着TNF-alpha的数量或持续时间,你会增加突变的数量,”Perli说。
“此外,我们想测试我们的系统。能够在小鼠中记录和提取来自小鼠的活细胞的信息可以帮助回答有意义的生物学问题,“崔说。研究人员表明,该系统能够在小鼠中记录炎症。
大多数突变导致缺失DNA序列的一部分,因此研究人员设计了其RNA引导股,比通常的20个核苷酸长,因此它们不会变得太短。40个核苷酸的序列足够长,以便记录一个月,并且研究人员还设计了70-核苷酸序列,其可用于记录生物信号甚至更长。
跟踪发展和疾病
研究人员还表明,通过在同一细胞中产生多个自定位RNA引导股,它们可以通过在同一细胞中产生多个自定位的RNA引导股来进行工程师来检测和记录多个输入。每个RNA引导件与特定输入连接,仅在存在该输入时产生。在这项研究中,研究人员表明,它们可以记录抗生素强霉素的存在和称为IPTG的分子。
研究人员表示,目前该方法最有可能用于研究人体细胞,组织或工程器官的研究。通过编程单元来记录多个事件,科学家可以使用该系统监测炎症或感染,或监测癌症进展。它对追踪细胞在从胚胎到成年人的动物的发展期间追踪细胞是如何专门化的。
“通过这种技术,您可以具有不同的内存寄存器,这些内存寄存器正在录制到不同的信号,并且您可以看到该单元中的每一个信号被小区接收到这种持续时间或在该强度上,”Perli说。“那种方式你可以更接近了解发展中发生的事情。”
出版物:Samuel D. Perli等,“与人类细胞中自定位Crisp-CA的连续遗传记录,”科学“,2016年8月18日; DOI:10.1126 / science.aag0511.
 健康教育网
健康教育网